La tubería de comandos Not-alike3 encuentra blancos genómicos para el diagnóstico molecular de enfermedades causadas por microorganismos patógenos
DOI:
https://doi.org/10.18633/biotecnia.v27.2495Palabras clave:
Programación Python, Diagnóstico Molecular, Detección de patógenos, Biología molecular, SHGResumen
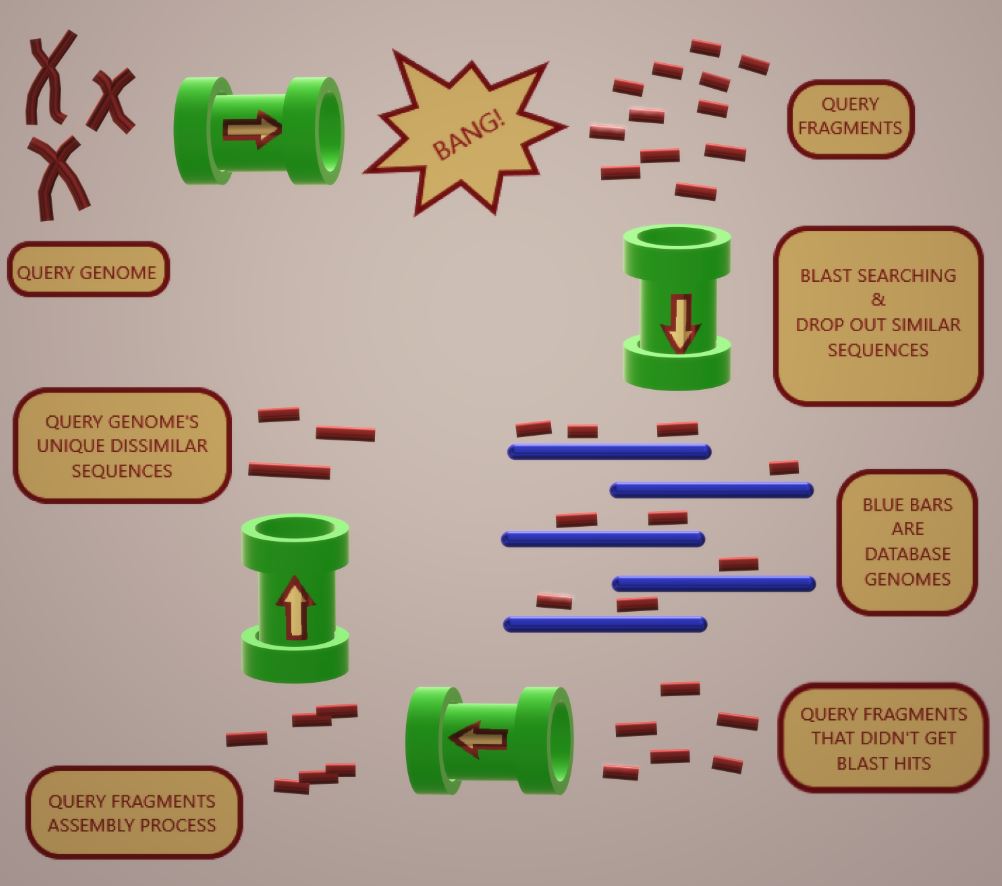
Las técnicas moleculares basadas en ADN son cruciales para la identificación correcta de microorganismos. A pesar de su velocidad y sensibilidad, la especificidad depende en el diseño de la sonda o el primer de PCR. La búsqueda por secuencias únicas en gnomas de organismos tiene varias aplicaciones como la misma identificación de microorganismos. Existen herramientas bioinformáticas publicadas que emplean el concepto de hibridación genómica sustractiva in silico para la identificación de regiones únicas o específicas de especie dentro de un genoma de interés. Sin embargo, éstas usan herramientas desactualizadas y/o lenguajes de programación que actualmente no son usados en el área de la bioinformática, otras herramientas son muy especializadas o son difíciles de obtener debido a que los repositorios (acervos) donde se hospedan las herramientas no son públicas o están apagados actualmente. Para abordar estos problemas, implementamos el concepto de hibridación genómica sustractiva in silico en una tubería de comandos escrita en Python que es amigable, de código abierto, gratis y fácil de instalar (llamada Not-alike3). Adicionalmente, diseñamos primers de PCR utilizando como molde la secuencia de regiones únicas identificadas en los genomas de dos especies de Mucorales, Rhizopus oryzae y Cuninghamella bertholletiae empleando la tubería Not-alike3, después realizamos evaluaciones de especificidad para desafiar esos primers que diseñamos y observamos que dichos primers fueron específicos de especie.
Descargas
Citas
Argimón, S., Konganti, K., Chen, H., Alekseyenko, A.V., Brown, S. and Caufield, P.W. 2014. Comparative genomics of oral isolates of Streptococcus mutans by in silico genome subtraction does not reveal accessory DNA associated with severe early childhood caries. Infection, Genetics and Evolution. 21: 269-278. DOI: https://doi.org/10.1016/j.meegid.2013.11.003.
Baldin, C., Soliman, S.S., Jeon, H.H., Alkhazraji, S., Gebremariam, T., Gu, Y., Bruno, V.M., Cor-nely, O.A., Leather, H.L., Sugrue, M.W., Wingard, J.R., Stevens, D.A., Edwards, J.E. and Ibra-him, A.S. 2018. PCR-based approach targeting mucorales-specific gene family for diagnosis of mucormycosis. Journal of clinical microbiology. 56(10): 1110-1128. DOI: https://doi.org/10.1128/jcm.00746-18.
Barh, D., Tiwari, S., Jain, N., Ali, A., Santos, A.R., Misra, A.N., Azevedo, V. and Kumar, A. 2011. In silico subtractive genomics for target identification in human bacterial pathogens. Drug Development Research. 72(2): 162-177. DOI: https://doi.org/10.1002/ddr.20413.
Camacho, C., Coulouris, G., Avagyan, V., Ma, N., Papadopoulos, J., Bealer, K. and Madden, T.L. 2009. BLAST+: architecture and applications. BMC bioinformatics. 10: 1-9. DOI: https://doi.org/10.1186/1471-2105-10-421.
Chetouani, F., Glaser, P. and Kunst, F. 2001. FindTarget: software for subtractive genome analy-sis. Microbiology. 147(10): 2643-2649. DOI: https://doi.org/10.1099/00221287-147-10-2643.
Davi, M.J.P., Jeronimo, S.M.B., Lima, J.P.M.S. and Lanza, D.C.F. 2021. 2021. Design and in silico validation of polymerase chain reaction primers to detect severe acute respiratory syndrome coro-navirus 2 (SARS-CoV-2). Scientific Reports. 11: 12565 https://doi.org/10.1038/s41598-021-91817-9.
Gardès, J., Croce, O. and Christen, R. 2012. In silico analyses of primers used to detect the patho-genicity genes of Vibrio cholerae. Microbes Environ. 27(3): 250-6. DOI: https://doi.org/10.1264/jsme2.me11317.
Haubold, B., Klötzl, F., Hellberg, L., Thompson, D. and Cavalar, M. 2021. Fur: Find unique ge-nomic regions for diagnostic PCR. Bioinformatics. 37(15): 2081-2087. doi: https://doi.org/10.1093/bioinformatics/btab059.
Kim, D., Paggi, J.M., Park, C., Bennett, C. and Salzberg, S.L. 2019. Graph-based genome align-ment and genotyping with HISAT2 and HISAT-genotype. Nature biotechnology. 37(8): 907-915. DOI: https://doi.org/10.1038/s41587-019-0201-4.
Li, H., Handsaker, B., Wysoker, A., Fennell, T., Ruan, J., Homer, N., Marth, G., Abecasis, G., Durbin, R. and 1000 Genome Project Data Processing Subgroup. 2009. The sequence align-ment/map format and SAMtools. Bioinformatics. 25(16): 2078-2079. DOI: https://doi.org/10.1093/bioinformatics/btp352.
Liu, Q., Jin, X., Cheng, J., Zhou, H., Zhang, Y. and Dai Y. 2023. Advances in the application of molecular diagnostic techniques for the detection of infectious disease pathogens (Review). Mol Med Rep. 27(5):104. DOI: https://doi.org/10.3892/mmr.2023.12991.
O’Leary, N.A., Cox, E., Holmes, J.B., Anderson, W.R., Falk, R., Hem, V., Tsuchiya, M.T.N., Schuler, G.D., Zhang, X., Torcivia, J., Ketter, A., Breen, L., Cothran, J., Bajwa, H., Tinne, J., Meric, P.A., Hlavina, W. and Schneider, V.A. 2024. Exploring and retrieving sequence and metadata for species across the tree of life with NCBI Datasets. Sci Data. 11(1): 732. DOI: https://doi.org/10.1038/s41597-024-03571-y
Pagès, H., Aboyoun, P., Gentleman, R. and DebRoy, S. 2024. Biostrings: Efficient manipulation of biological strings. Bioconductor R version package 2.70.2. DOI: https://doi.org/doi:10.18129/B9.bioc.Biostrings.
Pertea, G. and Pertea, M. 2020. GFF utilities: GffRead and GffCompare. F1000Research. 9. DOI: https://doi.org/10.12688%2Ff1000research.23297.2.
Pertea, M., Kim, D., Pertea, G.M., Leek, J.T. and Salzberg, S.L. 2016. Transcript-level expression analysis of RNA-seq experiments with HISAT, StringTie and Ballgown. Nature protocols. 11(9): 1650-1667. DOI: https://doi.org/10.1038/nprot.2016.095.
Portela, J., Grunau, C., Cosseau, C., Beltran, S., Dantec, C., Parrinello, H. and Boissier, J. 2010. Whole-genome in-silico subtractive hybridization (WISH)-using massive sequencing for the identi-fication of unique and repetitive sex-specific sequences: the example of Schistosoma mansoni. BMC genomics. 11: 1-8. DOI: https://doi.org/10.1186/1471-2164-11-387.
Shao, Y., He, X., Harrison, E.M., Tai, C., Ou, H.Y., Rajakumar, K. and Deng, Z. 2010. mGe-nomeSubtractor: a web-based tool for parallel in silico subtractive hybridization analysis of multiple bacterial genomes. Nucleic Acids Research. 38: W194-W200. DOI: https://doi.org/10.1093/nar/gkq326.
Singh, V. and Mishra, R.K. 2010. RISCI-Repeat Induced Sequence Changes Identifier: a com-prehensive, comparative genomics-based, in silico subtractive hybridization pipeline to identify re-peat induced sequence changes in closely related genomes. BMC bioinformatics. 11: 1-25. DOI: https://doi.org/10.1186/1471-2105-11-609.
Ueda, S., Washio, K. and Kurosaki, K. 1990. Human-specific sequences: Isolation of spe-cies-specific DNA regions by genome subtraction. Genomics. 8(1): 7-12. DOI: https://doi.org/10.1016/0888-7543(90)90219-K.
Untergasser, A., Cutcutache, I., Koressaar, T., Ye, J., Faircloth, B.C., Remm, M. and Rozen, S.G. 2012. Primer3 – New capabilities and interfaces, Nucleic Acids Research. 40(15): e115 – e115. DOI: https://doi.org/10.1093/nar/gks596.
van Weezep, E., Kooi, E.A. and van Rijn, P.A. 2019. PCR diagnostics: In silico validation by an automated tool using freely available software programs. J Virol. Methods. 270: 106-112. DOI: https://doi.org/10.1016/j.jviromet.2019.05.002.
Vidic, J., Manzano, M., Chang, C.M. and Jaffrezic-Renault, N. 2017. Advanced biosensors for detection of pathogens related to livestock and poultry. Veterinary Research. 48(1): 1-22. DOI: https://doi.org/10.1186/s13567-017-0418-5.
Voigt, K., Cigelnik, E. and O'donnell, K. 1999. Phylogeny and PCR identification of clinically important Zygomycetes based on nuclear ribosomal-DNA sequence data. Journal of Clinical Micro-biology. 37(12): 3957-3964. DOI: https://doi.org/10.1128/jcm.37.12.3957-3964.1999.
Wang, X., Fu, Y.F., Wang, R.Y., Li, L., Cao, Y.H., Chen, Y.Q., Zhao, H.Z., Zhang, Q.Q., Wu, J.Q., Weng, X.H., Cheng, X.J. and Zhu, L.P. 2014. Identification of clinically relevant fungi and prototheca species by rRNA gene sequencing and multilocus PCR coupled with electrospray ioni-zation mass spectrometry. PLoS One. 9(5): e98110. DOI: https://doi.org/10.1371/journal.pone.0098110.
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2025

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-CompartirIgual 4.0.
La revista Biotecnia se encuentra bajo la licencia Atribución-NoComercial-CompartirIgual 4.0 Internacional (CC BY-NC-SA 4.0)













_(2).jpg)








