Análisis del genoma de Bacillus paralicheniformis AA1 aislado de un sistema de milpa convencional en la región sur de Sonora, México
DOI:
https://doi.org/10.18633/biotecnia.v27.2552Palabras clave:
Bacillus paralicheniformis, genoma completo, agricultura sostenibleResumen
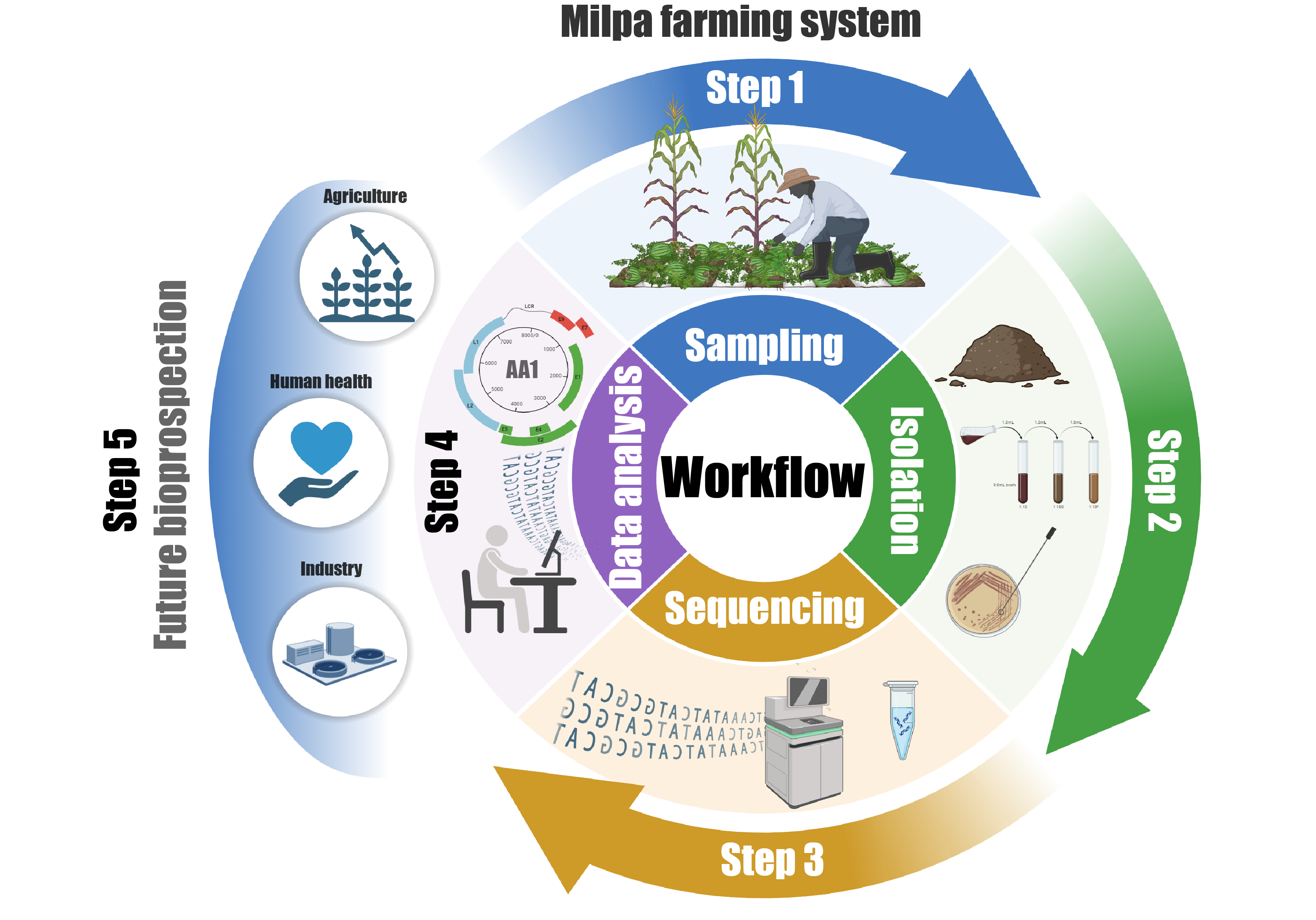
El presente estudio presenta un análisis genómico de Bacillus paralicheniformis AA1, una cepa bacteriana aislada de un sistema agrícola tradicional de milpa en Sonora, México. El análisis genómico reveló un alto nivel de integridad, evidenciado por la presencia de un repertorio diverso y funcionalmente significativo de genes asociados con procesos biológicos fundamentales, incluyendo la asimilación de nutrientes, la respuesta al estrés y la regulación celular. En particular, el genoma también contiene genes responsables de la biosíntesis de metabolitos secundarios, lo que resalta su potencial para
aplicaciones biotecnológicas. La clasificación taxonómica se
realizó rigurosamente mediante enfoques genómicos integrados, confirmando la identificación de la cepa AA1 como perteneciente a la especie B. paralicheniformis. El análisis genómico comparativo estableció además un alto grado de similitud genética entre B. paralicheniformis AA1 y otras cepas de B. paralicheniformis con capacidades biotecnológicas caracterizadas. Esta similitud sugiere que AA1 posee elementos genéticos responsables de la síntesis de compuestos antimicrobianos, enzimas de relevancia industrial y metabolitos que promueven el crecimiento vegetal. Los hallazgos resaltan el potencial de B. paralicheniformis AA1 como una cepa valiosa para la biotecnología y la agricultura sostenible. Este análisis contribuye al conocimiento necesario para el desarrollo de prácticas agrícolas innovadoras al mejorar nuestra comprensión de la diversidad microbiana en agroecosistemas tradicionales. Las investigaciones derivadas de este estudio deberán centrarse en la validación funcional de genes clave para comprender completamente el potencial de AA1 en la producción de antimicrobianos, la síntesis de enzimas y su aplicación en sistemas agrícolas sostenibles.
Descargas
Citas
Delgado-Baquerizo M, Oliverio AM, Brewer TE, Benavent-González A, Eldridge DJ, Bardgett RD, Maestre FT, Singh BK, Fierer N. 2018. A global atlas of the dominant bacteria found in soil. Science (1979) 359:320–325.
Steinke K, Mohite OS, Weber T, Kovács ÁT. 2021. Phylogenetic Distribution of Secondary Metabolites in the Bacillus subtilis Species Complex. mSystems 6.
Su Y, Liu C, Fang H, Zhang D. 2020. Bacillus subtilis: a universal cell factory for industry, agriculture, biomaterials and medicine. Microbial Cell Factories 2020 19:1 19:1–12.
Iqbal S, Qasim M, Rahman H, Khan N, Paracha RZ, Bhatti MF, Javed A, Janjua HA. 2023. Genome mining, antimicrobial and plant growth-promoting potentials of halotolerant Bacillus paralicheniformis ES-1 isolated from salt mine. Molecular Genetics and Genomics 298:79–93.
Ashajyothi M, Mahadevakumar S, Venkatesh YN, Sarma PVSRN, Danteswari C, Balamurugan A, Prakash G, Khandelwal V, Tarasatyavathi C, Podile AR, Kirankumar MS, Chandranayaka S. 2024. Comprehensive genomic analysis of Bacillus subtilis and Bacillus paralicheniformis associated with the pearl millet panicle reveals their antimicrobial potential against important plant pathogens. BMC Plant Biol 24:1–23.
Swietczak J, Kalwasińska A, Felföldi T, Swiontek Brzezinska M. 2023. Bacillus paralicheniformis 2R5 and its impact on canola growth and N-cycle genes in the rhizosphere. FEMS Microbiol Ecol 99.
Chavarria-Quicaño E, Contreras-Jácquez V, Carrillo-Fasio A, De la Torre-González F, Asaff-Torres A. 2023. Native Bacillus paralicheniformis isolate as a potential agent for phytopathogenic nematodes control. Front Microbiol 14.
Jinal HN, Sakthivel K, Amaresan N. 2020. Characterisation of antagonistic Bacillus paralicheniformis (strain EAL) by LC–MS, antimicrobial peptide genes, and ISR determinants. Antonie van Leeuwenhoek, International Journal of General and Molecular Microbiology 113:1167–1177.
Díaz-Manzano FE, Amora DX, Martínez-Gómez Á, Moelbak L, Escobar C. 2023. Biocontrol of Meloidogyne spp. in Solanum lycopersicum using a dual combination of Bacillus strains. Front Plant Sci 13:1077062.
Hemangini BD, Kiran CS, Vrinda TS, Bhatt Hemangini CD. 2018. Antimicrobial activity of Bacillus paralicheniformis SUBG0010 against plant pathogenic bacteria of mango. ~ 449 ~ Journal of Pharmacognosy and Phytochemistry 7:449–455.
Pawaskar M. 2023. In-vivo biocontrol of Fusarium wilt in chilli plants using hypersaline Bacillus paralicheniformis strain MPSK23 https://doi.org/10.21203/RS.3.RS-2927693/V1.
Fonteyne S, Castillo Caamal JB, Lopez-Ridaura S, Van Loon J, Espidio Balbuena J, Osorio Alcalá L, Martínez Hernández F, Odjo S, Verhulst N. 2023. Review of agronomic research on the milpa, the traditional polyculture system of Mesoamerica. Frontiers in Agronomy 5:1115490.
Compant S, Samad A, Faist H, Sessitsch A. 2019. A review on the plant microbiome: Ecology, functions, and emerging trends in microbial application. J Adv Res 19:29–37.
Bankevich A, Nurk S, Antipov D, Gurevich AA, Dvorkin M, Kulikov AS, Lesin VM, Nikolenko SI, Pham S, Prjibelski AD, Pyshkin A V., Sirotkin A V., Vyahhi N, Tesler G, Alekseyev MA, Pevzner PA. 2012. SPAdes: A New Genome Assembly Algorithm and Its Applications to Single-Cell Sequencing. Journal of Computational Biology 19:455.
Tatusova T, Dicuccio M, Badretdin A, Chetvernin V, Nawrocki EP, Zaslavsky L, Lomsadze A, Pruitt KD, Borodovsky M, Ostell J. 2016. NCBI prokaryotic genome annotation pipeline. Nucleic Acids Res 44:6614–6624.
Eren AM, Kiefl E, Shaiber A, Veseli I, Miller SE, Schechter MS, Fink I, Pan JN, Yousef M, Fogarty EC, Trigodet F, Watson AR, Esen ÖC, Moore RM, Clayssen Q, Lee MD, Kivenson V, Graham ED, Merrill BD, Karkman A, Blankenberg D, Eppley JM, Sjödin A, Scott JJ, Vázquez-Campos X, McKay LJ, McDaniel EA, Stevens SLR, Anderson RE, Fuessel J, Fernandez-Guerra A, Maignien L, Delmont TO, Willis AD. 2020. Community-led, integrated, reproducible multi-omics with anvi’o. Nature Microbiology 2020 6:1 6:3–6.
Lee MD. 2019. GToTree: a user-friendly workflow for phylogenomics. Bioinformatics 35:4162–4164.
Grant JR, Enns E, Marinier E, Mandal A, Herman EK, Chen CY, Graham M, Van Domselaar G, Stothard P. 2023. Proksee: in-depth characterization and visualization of bacterial genomes. Nucleic Acids Res 51:W484–W492.
Klimke W, O’Donovan C, White O, Brister JR, Clark K, Fedorov B, Mizrachi I, Pruitt KD, Tatusova T. 2011. Solving the Problem: Genome Annotation Standards before the Data Deluge. Stand Genomic Sci 5:168.
Berais-Rubio A, Morel-Revetria M, Filippi CV, Reyno R, Monza J. 2023. Ensifer meliloti elite strain U143 used as alfalfa inoculant in Uruguay: Characterization and draft genome sequence. The Microbe 1:100008.
Berriel V, Morel MA, Filippi C V., Monza J. 2021. Draft genome sequence of Bradyrhizobium sp. strain Oc8 isolated from Crotalaria ochroleuca nodule. Curr Res Microb Sci 2:100074.
Kim M, Chun J. 2014. 16S rRNA Gene-Based Identification of Bacteria and Archaea using the EzTaxon Server. Methods in Microbiology 41:61–74.
Bars-Cortina D, Moratalla-Navarro F, García-Serrano A, Mach N, Riobó-Mayo L, Vea-Barbany J, Rius-Sansalvador B, Murcia S, Obón-Santacana M, Moreno V. 2023. Improving Species Level-taxonomic Assignment from 16S rRNA Sequencing Technologies. Curr Protoc 3:e930.
Parks DH, Chuvochina M, Rinke C, Mussig AJ, Chaumeil PA, Hugenholtz P. 2022. GTDB: an ongoing census of bacterial and archaeal diversity through a phylogenetically consistent, rank normalized and complete genome-based taxonomy. Nucleic Acids Res 50:D785–D794.
Arahal DR. 2014. Whole-Genome Analyses: Average Nucleotide Identity. Methods in Microbiology 41:103–122.
Asif M, Li-Qun Z, Zeng Q, Atiq M, Ahmad K, Tariq A, Al-Ansari N, Blom J, Fenske L, Alodaini HA, Hatamleh AA. 2023. Comprehensive genomic analysis of Bacillus paralicheniformis strain BP9, pan-genomic and genetic basis of biocontrol mechanism. Comput Struct Biotechnol J 21:4647–4662.
Annapurna K, Govindasamy V, Sharma M, Ghosh A, Chikara SK. 2018. Whole genome shotgun sequence of Bacillus paralicheniformis strain KMS 80, a rhizobacterial endophyte isolated from rice (Oryza sativa L.). 3 Biotech 8:1–4.
Valenzuela-Ruiz V, Robles-Montoya RI, Parra-Cota FI, Santoyo G, del Carmen Orozco-Mosqueda M, Rodríguez-Ramírez R, de los Santos-Villalobos S. 2019. Draft genome sequence of Bacillus paralicheniformis TRQ65, a biological control agent and plant growth-promoting bacterium isolated from wheat (Triticum turgidum subsp. durum) rhizosphere in the Yaqui Valley, Mexico. 3 Biotech 9:1–7.
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2025

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-CompartirIgual 4.0.
La revista Biotecnia se encuentra bajo la licencia Atribución-NoComercial-CompartirIgual 4.0 Internacional (CC BY-NC-SA 4.0)













_(2).jpg)








